High Resolution Melt
L'analyse des courbes de fusion à haute résolution, ou High Resolution Melt (HRM) est une technique de biologie moléculaire permettant la détection de mutations, de polymorphisme génétique et de différences épigénétiques dans des échantillons d'ADN double brin. Elle a été découverte et développée par Idaho Technology à l'Université d'Utah[1]. Elle présente divers avantages par rapport aux autres techniques de génotypage :
- L'HRM coûte moins cher que d'autres techniques comme le séquençage et le Taqman. Cela en fait une technique de choix pour les projets à grande échelle.
- Elle est relativement rapide et simple.
Méthode[modifier | modifier le code]
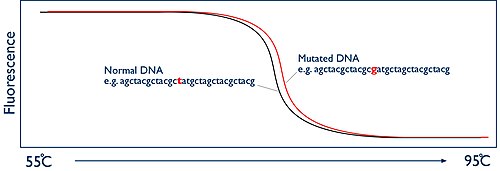
L'analyse HRM est réalisée à partir d'échantillons d'ADN double brin. La région de l'ADN potentiellement porteuse de la mutation est d'abord amplifiée par PCR. L'amplicon est alors chauffé très progressivement et précisément d'environ 50 à 95 °C. À un certain point, les deux brins d'ADN se séparent : c'est ce processus que l'on appelle fusion de l'ADN. Le principe de la HRM est d'observer cette fusion en temps réel.
Cela est possible grâce à des composés particuliers, appelés colorants intercalants, qui ont la propriété de devenir plus fluorescents lorsqu'ils sont fixés à de l'ADN double brin. En l'absence de double brin, ils ne peuvent pas se fixer et leur niveau de fluorescence est très faible. Au début de l'analyse HRM, à la plus basse température, l'intensité de fluorescence est très importante, car l'ensemble de l'amplicon est présent sous forme double brin. Mais, au fur et à mesure que la température augmente et que les brins se séparent, la fluorescence diminue. L'enregistrement de l'intensité de fluorescence en fonction de la température est appelée courbe de fusion.
Un changement, même mineur, dans la séquence de l'ADN conduit à une variation de la cinétique de fusion, qui peut être détectée grâce à la haute résolution utilisée.
Type sauvage, hétérozygote ou homozygote[modifier | modifier le code]
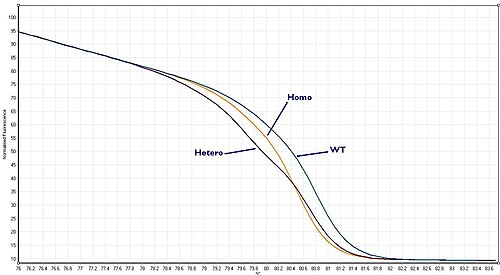
Comme un sujet possède deux copies de chaque gène, il y a trois possibilités :
- Aucun allèle ne contient la mutation (Type sauvage)
- L'un ou l'autre des allèles contient la mutation (Hétérozygote)
- Les deux allèles contiennent la mutation (Homozygote)
Les deux allèles étant amplifiés simultanément, la courbe de fluorescence que l'on observe correspond à la somme des courbes de fluorescences des deux allèles. Si la résolution est suffisante, il est possible de les distinguer :
Applications de la HRM[modifier | modifier le code]
Génotypage de SNP[modifier | modifier le code]
Les SNP (single nucleotide polymorphism) sont des mutations n'affectant qu'un nucléotide. Il est possible de les détecter par HRM, ce qui est bien moins coûteux que l'utilisation d'une puce à ADN. Par exemple, la substitution d'une guanine par une adénine sur le gène Vssc (Voltage sensitive sodium channel) confère une résistance à la permethrine, un acaricide, chez l'acarien Scabies (responsable de la gale). Ce SNP entraîne une température de fusion légèrement plus basse pour l'ADN des acariens résistants. Cela est dû au fait que la liaison G-C est plus forte que la liaison A-T, en raison du nombre de liaisons hydrogène impliquées[2].
En diagnostic clinique, la HRM est parfois préférée ou associée au séquençage pour identifier des mutations[3],[4] (par exemple, plus de 400 mutations ont été identifiées sur les gènes BRCA1 et BRCA2, impliqués dans le cancer du sein). Actuellement, la HRM est employée principalement comme outil de screening, et selon le besoin complétée par un séquençage.
Test de zygosité[modifier | modifier le code]
Le test de zygosité permet de savoir si deux personnes sont de « vrais » jumeaux ou des jumeaux fraternels. Parmi les nombreuses méthodes utilisables, l'HRM peut être envisagée.
Épigénétique[modifier | modifier le code]
La méthodologie HRM a aussi été utilisée pour analyser le statut de méthylation de l'ADN. Cela a une importance particulière en oncogénétique car la méthylation de l'ADN peut diminuer l'expression des gènes régulateurs des tumeurs, comme ceux qui régulent l'apoptose et la réparation de l'ADN. Elle a aussi une influence sur la réponse aux chimiothérapies. Par exemple, si le promoteur du gène de réparation MGMT (O-6-méthylguanine-DNA méthyltransférase) est méthylé, la tumeur sera moins sensible aux agents alkylants. Lors d'une étude[5] conduite par HRM, sur 19 cancers colorectaux analysés, 8 présentaient une méthylation du promoteur MGMT.
Pour obtenir ce résultat, l'échantillon d'ADN est traité par le bisulfite, qui a la propriété de transformer les cytosines non-méthylées (et uniquement celles-là) en uracile. Ainsi, la séquence de bases est modifiée, ce qui affecte la courbe de fusion et peut être détecté lors de l'analyse HRM. La HRM permet de connaître la proportion de méthylation dans un échantillon donné, en comparant la courbe de fusion à une gamme progressive réalisée à partir de mélanges d'ADN non-méthylé et d'ADN entièrement méthylés.
Colorants intercalants employés[modifier | modifier le code]
Le premier colorant à avoir été employé en HRM est le SYBR Green. Il ne fluoresce que lorsqu'il est lié à de l'ADN double-brin. À hautes concentrations, il peut inhiber la réaction de polymérisation lors de la PCR. Son utilisation est possible à basse concentration, mais déconseillée par certains chercheurs[6], en raison du phénomène de dye jumping : des molécules de colorant libérées par la fusion en certains points sont réincorporés à un autre endroit où les brins ne sont pas encore séparés, conduisant à une erreur sur les résultats[7],[6]. De nouveaux colorants saturants comme le LC Green, le LC Green Plus, le ResoLight, l'EvaGreen, le Chromofy et le SYTO 9 sont disponibles sur le marchés et ont montré leur efficacité pour la HRM. Cependant, le SYBR Green est toujours employé avec certains appareils[8].
Références[modifier | modifier le code]
- Pour un historique de l'HRM, voir http://www.dna.utah.edu/Hi-Res/TOP_Hi-Res%20Melting.html
- Pasay C, Arlian L, Morgan M, et al., « High-resolution melt analysis for the detection of a mutation associated with permethrin resistance in a population of scabies mites », Med. Vet. Entomol., vol. 22, no 1, , p. 82–8 (PMID 18380658, DOI 10.1111/j.1365-2915.2008.00716.x, lire en ligne)
- James PA, Doherty R, Harris M, et al., « Optimal selection of individuals for BRCA mutation testing: a comparison of available methods », J. Clin. Oncol., vol. 24, no 4, , p. 707–15 (PMID 16446345, DOI 10.1200/JCO.2005.01.9737, lire en ligne)
- Krypuy M, Ahmed AA, Etemadmoghadam D, et al., « High resolution melting for mutation scanning of TP53 exons 5-8 », BMC Cancer, vol. 7, , p. 168 (PMID 17764544, PMCID 2025602, DOI 10.1186/1471-2407-7-168, lire en ligne)
- Wojdacz TK, Dobrovic A, « Methylation-sensitive high resolution melting (MS-HRM): a new approach for sensitive and high-throughput assessment of methylation », Nucleic Acids Res., vol. 35, no 6, , e41 (PMID 17289753, PMCID 1874596, DOI 10.1093/nar/gkm013, lire en ligne)
- Reed GH, Kent JO, Wittwer CT, « High-resolution DNA melting analysis for simple and efficient molecular diagnostics », Pharmacogenomics, vol. 8, no 6, , p. 597–608 (PMID 17559349, DOI 10.2217/14622416.8.6.597, lire en ligne) as PDF
- Gundry CN, Vandersteen JG, Reed GH, Pryor RJ, Chen J, Wittwer CT, « Amplicon melting analysis with labeled primers: a closed-tube method for differentiating homozygotes and heterozygotes », Clin. Chem., vol. 49, no 3, , p. 396–406 (PMID 12600951, DOI 10.1373/49.3.396, lire en ligne)
- Pornprasert S, Phusua A, Suanta S, Saetung R, Sanguansermsri T, « Detection of alpha-thalassemia-1 Southeast Asian type using real-time gap-PCR with SYBR Green1 and high resolution melting analysis », Eur. J. Haematol., vol. 80, no 6, , p. 510–4 (PMID 18284625, DOI 10.1111/j.1600-0609.2008.01055.x, lire en ligne)
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « High Resolution Melt » (voir la liste des auteurs).