Utilisateur:Lewisiscrazy/Cinétique enzymatique

La cinétique enzymatique a pour objet d'identifier et de décrire les mécanismes des réactions biochimiques, catalysées par les enzymes (réaction enzymatique), en étudiant leur vitesse c'est-à-dire leur évolution en fonction du temps. Ce genre d'étude peut révéler le mécanisme catalytique, donner des indications sur son rôle dans le métabolisme, sur la façon dont son activité est contrôlée, et sur le mode d'action d'inhibiteurs. La cinétique enzymatique a donc de nombreuses applications dans les industries chimique et agroalimentaire ainsi que dans les biotechnologies et la médecine (pharmacologie, toxicologie).
Les enzymes sont généralement des protéines. Elles jouent le rôle de catalyseur dans des réactions chimiques qui transforment une molécule cible (qu'on appelle le substrat de l'enzyme) en des produits de la réaction. Le substrat se lie à une partie de l'enzyme appelée le site actif et subit une série d'étapes qu'on appelle le mécanisme enzymatique
- E + S ⇄ ES ⇄ ES* ⇄ EP ⇄ E + P
On distingue habituellement les réactions qui transforment un substrat unique et les réactions qui font intervenir plusieurs substrats.
La dihydrofolate réductase est un exemple d'enzyme qui transforme plusieurs substrats et plusieurs produits. Dans ce cas, les études cinétiques peuvent permettre de déterminer dans quel ordre les substrats se lient et dans quel ordre les produits sont libérés enzymatiques peuvent également montrer l'ordre dans lequel les substrats de lier et de la séquence dans laquelle les produits sont libérés. Un exemple d'enzyme qui transforme un substrat unique en plusieurs produits est la protéase, qui casse des laisons peptidiques pour former deux peptides à partir d'une protéine plus grosse. Au contraire, l'enzyme ADN polymérase combine deux nucléotides. Bien que ces mécanismes catalytiques consistent en une série complexe d'étapes, il arrive que l'une d'entre elles soit plus lente que toutes les autres: elle détermine alors la vitesse globale, on parle d'étape limitante en vitesse (ou d'étape déterminante en vitesse). Il peut s'agir d'une réaction chimique, d'un changement de conformation de l'enzyme etc.
La connaissance de la structure tridimensionnelle de l'enzyme est utile pour interpréter les données cinétiques. Par exemple, la structure peut suggérer la façon dont les substrats et les produits se lient au cours du cycle catalytique; quels changements de conformation se produisent au cours de la réaction; quel est le rôle particulier des chaînes latérales de certains résidus (les acides aminés qui composent l'enzyme). Si l'enzyme change significativement de conformation pendant le cycle catalytique, il peut être utile de déterminer ses structures en présence et en l'absence d'un analogue du substrat (une molécule qui a une structure voisine de celle du substrat, mais suffisamment différente pour ne pas être transformée par l'enzyme).
Certains catalyseurs biologiques ne sont pas des protéines: les ribozymes et les ribosomes sont des catalyseurs à base d'ARN essentiels pour de nombreuses fonctions cellulaires, telles que l'épissage de l'ARN et la traduction. La principale différence entre les ribozymes et les enzymes est que les catalyseurs à ARN sont composés de nucléotides, alors que les enzymes sont constituées de protéines (d'acides aminés). Mais les mécanismes réactionnels peuvent être étudiés de la même façon dans les deux cas.
Principes généraux[modifier | modifier le code]
L'équation bilan de la réaction catalysée par une enzyme est la même que celle de la réaction non catalysée: même réactifs et même produits. Comme l'enzyme est un catalyseur, elle ne modifie pas la position de l'équilibre de la réaction qui convertit les substrats et produits.[1] Cependant, la présence de l'enzyme augmente la vitesse de la réaction. Cette vitesse suit une cinétique très particulière: à basse concentration en substrat, la vitesse de la réaction est proportionnelle à la concentration en substrat; à très haute concentration en substrat la vitesse tend assymtotiquement vers un plateau (on dit qu'elle sature, ou qu'elle atteint sa valeur maximale). La raison de ce comportement est qu'à basse concentration en substrat, l'enzyme est en solution essentiellement sous une forme "libre", non liée au substrat; alors que dans la limite des hautes concentrations en substrat, toute les molécules d'enzymes en solution sont sous une forme complexée au substrat et la vitesse de la réaction devient déterminée par la fréquence à laquelle le complexe enzyme-substrat peut former le produit: cette fréquence est appelée constante catalytique, souvent notée <math>k_{\rm cat}</math>.[2] La concentration en substrat à laquelle la vitesse est la moitié de la vitesse maximale est appelée constante de Michaelis, notée KM
Les propriétés cinétiques les plus importantes de l'enzyme sont donc la fréquence intrinsèque de catalyse k_{\rm cat}, la facilité avec laquelle l'enzyme lie le substrat (mesurée par la valeur de Km), et le rapport kcat/KM, qui mesure la spécificité de l'enzyme pour un substrat particulier, pour des raisons détaillées plus bas. Connaitre les valeurs de ces paramètres peut aider à comprendre le rôle joué par une enzyme in vivo.
Les mes sures d'activités ("essais enzymatiques", en anglais: enzyme assays).[modifier | modifier le code]

Les essais enzymatiques sont des procédures de laboratoire qui permettent de mesurer la vitesse des réactions enzymatiques. Comme pour toute mesure de vitesse d'une réaction, il s'agit de mesurer la vitesse avec laquelle une concentration en substrat ou produit varie au cours du temps (la concentration de l'enzyme, elle, ne change pas, puisqu'il s'agit d'un catalyseur). Les méthodes de mesures sont aussi variées que les substrats et produits des réactions enzymatiques. On peut utiliser des méthodes spectrophotométriques si les réactifs et produits absorbent ou émettent de la lumière à des longueurs d'onde différentes; des mesures radiométriques ou de spectrométrie de masse peuvent permettent de détecter l'incorporation ou la libération d'éléments radioactifs. On préfèrera une technique qui permet de suivre la réaction en continu, plutôt qu'une technique qui requiert que l'on interrompe la réaction à un temps donné pour doser la concentration des réactifs restants ou des produits formés.[3]
Les essais enzymatiques les plus sensibles permettent d'utiliser un faisseau laser pour mesurer des changements de fluoresence de molécules d'enzymes isolées.[4] Ces techniques complémentent les approches traditionnelles en cinétique enzymatique, qui consistent à quantifier le comportement moyen des populations de millions de molécules d'enzyme.[5][6]
La figure ci contre est un exemple de courbe de progression pour une réaction enzymatique. Pendant une certaine période après le début de la réaction, ll'enzyme transforme le substrat à une vitesse qui constante (la concentration en produit varie linéairement avec le temps). Comme la réaction consomme le substrat et fait décroître sa concentration, la vitesse diminue continuement. La durée de cette période (typiquement, des minutes ou dizaines de minutes) dépend beaucoup du système qui est étudié. L'approche classique en cinétique enzymatique consiste à répéter des mesures de vitesses initiales dans des expériences distinctes, dans lesquelles on utilise des concentrations initiales en substrat différentes.
Cependant, on peut aussi s'intéresser alternativement à l'évolution du système sur des temps très courts ou très longs. Certains équipements permettent de détecter des variations de concentrations sur des temps très courts (moins d'une seconde). Ces mesures d'analyse rapides sont essentielles pour les mesures "pré-stationnaires" discutées ci dessous. Il est également possible de mesurer la variation complète des concentration jusqu'à la fin de la réaction, quand elle s'arrête (soit que tous les réactifs sont consommés, soit que l'équilibre est atteint), et analyser la forme de ce qu'on appelle la courbe de progrès.[7] Cette approche est utile quand la vitesse initiale est trop rapide pour être mesurée avec précision.
Cinétique Michaélienne[modifier | modifier le code]
Les enzymes qui transforment une molécule de substrat suivent souvent une cinétique dite "Michaelienne". Des exemples de telles enzyles sont l'isomérase comme la triosephosphateisomerase ou la bisphosphoglycerate mutase, les lyases intramoléculaires comme l'adénylate cyclase et le ribozyme.[8] Toutefois, certaines enzymes, qui transforment un substrat unique suivent es cinétiques différentes. La Catalase en est un exemple: cette enzyme réagit avec une première molécule de peroxyde d'hydrogène qu'elle oxyde, puis par une deuxième molécule qu'elle réduit; un seul type de substrat est concerné mais l'équation bilan fait intervenir deux molécules de substrat et le mécanisme de la catalase est en fait un mécanisme de type ping–pong, décrit ci dessous dans la partie sur les enzymes à plusieurs substrats.
L'équation de Michaelis et Menten
Les vitesses des réactions catalysées par les enzymes ne varient pas de façon linéaire avec la concentration en substrat. Comme dit plus haut, la vitesse de la réaction atteint une valeur limite maximale à concentration en substrat infinie.
Le modèle cinétique classique pour catalyse enzymatique de trasnformation d'une seule molécule de substrat en produit est indiquée sur la droite. Il s'agit d'une réaction bimoléculaire entre l'enzyme E et le substrat S pour former le complexe enzyme–substrat ES, suivi d'une étape irréversible de formation du produit P et de régénération de l'enzyme libre E. Le taux de réaction enzymatique augmente avec l'augmentation de la concentration en substrat jusqu'à un certain niveau, appelé Vmax; au Vmax, l'augmentation de la concentration du substrat ne cause pas d'augmentation de la vitesse de réaction comme il n'y avait plus d'enzyme (E) disponible pour réagir avec le substrat (S). Ici, la vitesse de la réaction dépend de la ES complexe et la réaction devient une réaction unimoléculaire avec un ordre de zéro. Bien que le mécanisme enzymatique de la réaction unimoléculaire
peut être assez complexe, il y a généralement un taux de détermination enzymatique étape qui permet cette réaction à être modélisé comme une simple étape catalytique avec une apparente unimoléculaire constante de vitesse kcat. Si la réaction chemin de cession par rapport à un ou plusieurs intermédiaires, kcat sera fonction de plusieurs élémentaire des constantes de vitesse, alors que, dans le cas le plus simple d'une unique élémentaire de la réaction (par exemple, pas d'intermédiaires) il sera identique à la primaire unimoléculaire constante de vitesse k2. L'apparente unimoléculaire constante de vitesse kde chat est aussi appelé nombre de roulement et indique le nombre maximum de réactions enzymatiques catalysées par seconde.
L'équation de Michaelis–Menten décrit comment la vitesse initiale de la réaction dépend de v0 dépend de la position de la liaison du substrat de l'équilibre et de la constante de vitesse k2.
Cette équation de Michaelis–Menten est la base pour la plupart un seul substrat de l'enzyme de cinétique. Deux hypothèses sous-tendent cette équation (en dehors de l'hypothèse générale sur le mécanisme ne comportant pas d'intermédiaire ou de l'inhibition de produits, et il n'y a pas de allostericity ou coopérativité). La première hypothèse est la soi-disant quasi-stable à l'état d'hypothèse (ou pseudo-état stable hypothèse), à savoir que la concentration de la lié au substrat de l'enzyme (et donc aussi la unbound modifications enzymatiques) beaucoup plus lentement que celles du produit et le substrat et ainsi, le changement dans le temps de l'immeuble peut être mis à zéro . La deuxième hypothèse est que le total de la concentration de l'enzyme ne change pas au fil du temps, ainsi . Une dérivation complet peut être trouvé ici.
La constante de Michaelis KM est expérimentalement définie comme la concentration à laquelle le taux de la réaction enzymatique est la moitié de Vmax, qui peut être vérifié par la substitution de [S] = Km dans l'équation de Michaelis–Menten et peut également être vu sous forme de graphique. Si le taux de détermination enzymatique étape est lent par rapport au substrat de dissociation (), la constante de Michaelis KM est à peu près la constante de dissociation KD des ES complexe.
Si
est petite par rapport à ensuite, le terme et de plus, très peu de ES complexe est formé, ainsi . Par conséquent, le taux de formation du produit est
Ainsi, la formation du produit taux dépend de la concentration de l'enzyme ainsi que sur la concentration en substrat, l'équation ressemble à une réaction bimoléculaire, avec une pseudo-constante de vitesse de second ordre . Cette constante est une mesure de l' efficacité catalytique. Le plus efficace des enzymes de parvenir à un dans la gamme de 108 – 1010 M−1 s−1. Ces enzymes sont tellement efficaces, elles permettent de catalyser une réaction à chaque fois qu'ils rencontrent une molécule de substrat et ont ainsi atteint une limite supérieure limite théorique l'efficacité (diffusion de la limite), et sont parfois appelés cinétiquement parfait enzymes.[9], Mais la plupart des enzymes sont loin d'être parfait: les valeurs moyennes de et sont sur le et , respectivement.[10]
Linéarisation de l'équation de Michaelis–Menten[modifier | modifier le code]
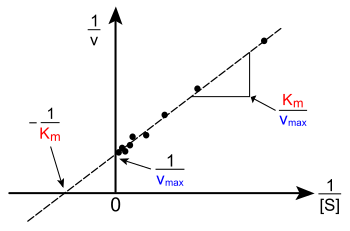
L'intrigue de v contre [S] ci-dessus n'est pas linéaire, bien qu'initialement linéaire à basse [S], il se penche pour saturer à haute [S]. Avant l'ère moderne de la non linéaire de la courbe d'ajustement sur les ordinateurs, de cette non-linéarité pourrait rendre difficile l'estimation de K,M et Vmax avec précision. Par conséquent, plusieurs chercheurs ont développé des linéarisations de l'équation de Michaelis–Menten, comme le Lineweaver–Burk de la parcelle, la Eadie–t. p. hofstee diagramme et le Hanes–Woolf parcelle. L'ensemble de ces représentations linéaires peut être utile pour la visualisation de données, mais aucun ne doit être utilisé pour déterminer les paramètres cinétiques, comme le logiciel informatique est facilement disponible qui permet une détermination plus précise par régression non linéaire méthodes.[11]
Le Lineweaver–Burk de la parcelle ou double réciproque de la parcelle est une commune de la manière d'illustrer des données cinétiques. C'est produit par la prise de la réciprocité des deux côtés de l'équation de Michaelis–Menten. Comme illustré sur la droite, c'est une forme linéaire de l'équation de Michaelis–Menten et produit une ligne droite avec l'équation y = mx + c avec un y-intercept équivalent à 1/Vmax et un xd'intersection du graphe représentant -1/KM.
Naturellement, pas de les valeurs expérimentales peuvent être prises au négatif de 1/[S]; la limite inférieure de la valeur de 1/[S] = 0 ( y-intercept) correspond à une infinie concentration en substrat, où 1/v=1/Vmax , comme illustré à droite; ainsi, le x-intercept est une extrapolation des données expérimentales prises à des concentrations positives. Plus généralement, la Lineweaver–Burk de la parcelle fausse l'importance des mesures prises à de faibles concentrations de substrat et, par conséquent, peuvent produire des estimations inexactes de Vmax et KM.[12] plus précis linéaire méthode de traçage est le Eadie-t. p. hofstee de la parcelle. Dans ce cas, v est tracée en fonction de v/[S]. Dans la troisième commune de la représentation linéaire, la Hanes-Woolf parcelle, "[S]/v est tracée en fonction de [S]. En général, les données de la normalisation peut aider à réduire la quantité de travail expérimental et peut augmenter la fiabilité de la sortie, et est adapté pour les graphiques et l'analyse numérique.[13]
La portée pratique de constantes cinétiques[modifier | modifier le code]
L'étude de la cinétique enzymatique est important pour deux raisons essentielles. Tout d'abord, elle permet d'expliquer comment les enzymes de travail, et d'autre part, il permet de prédire comment les enzymes se comporter dans les organismes vivants. Les constantes cinétiques défini ci-dessus, K,M et Vmax, sont essentielles pour la tente de comprendre comment les enzymes travaillent ensemble pour contrôler le métabolisme.
Faire ces prédictions n'est pas anodin, même pour les systèmes simples. Par exemple, l'oxaloacétate est formé par la malate déshydrogénase à l'intérieur de la mitochondrie. Oxaloacétate peut ensuite être consommé par la citrate synthase, la phosphoénolpyruvate carboxykinase ou de l'aspartate aminotransférase, de l'alimentation dans le cycle d'acide citrique, de la néoglucogenèse ou de l'acide aspartique biosynthèse, respectivement. Être capable de prédire combien oxaloacétate va dans le cheminement qui nécessite la connaissance de la concentration de l'oxaloacétate ainsi que la concentration et de la cinétique de chacune de ces enzymes. Ce but de prédire le comportement des voies métaboliques atteint sa forme la plus complexe de l'expression dans la synthèse d'énormes quantités de cinétique et de l'expression des gènes des données dans les modèles mathématiques de l'ensemble des organismes. Sinon, une simplification utile du métabolisme et de la modélisation du problème est d'ignorer le sous-jacent de la cinétique enzymatique et seulement s'appuyer sur des informations à propos de la réaction du réseau de la stoechiométrie, une technique appelée analyse du bilan flux.[14][15]
Cinétique de Michaelis–Menten avec l'intermédiaire[modifier | modifier le code]
On pourrait aussi envisager le moins de cas simple
où un complexe avec l'enzyme et un intermédiaire existe et que l'intermédiaire est converti en produit dans une deuxième étape. Dans ce cas, nous avons une très similaires équation[16]
Multi-substrat réactions[modifier | modifier le code]
Multi-substrat réactions suivre complexes de taux d'équations qui décrivent la manière dont les substrats de lier et dans quel ordre. L'analyse de ces réactions est beaucoup plus simple si la concentration de substrat est maintenue constante et le substrat B variées. Dans ces conditions, l'enzyme se comporte comme un simple substrat de l'enzyme et un terrain de v [S] donne apparente KM et Vmax constantes pour substrat B. Si un ensemble de ces mesures est effectuée à différentes fixe les concentrations de A, ces données peuvent être utilisées pour travailler ce que le mécanisme de la réaction est de. Pour une enzyme qui prend deux substrats A et B et les transforme en deux produits P et Q, il existe deux types de mécanisme: complexe ternaire et une table de ping–pong.
Mécanisme impliquant un complexe ternaire[modifier | modifier le code]
Dans ces enzymes, les deux substrats de se lier à l'enzyme dans le même temps à produire un VAE complexe ternaire. L'ordre de liaison peut être aléatoire (au hasard mécanisme) ou les substrats ont à se lier à une séquence particulière (dans un ordre mécanisme). Lorsqu'un ensemble de v [S] courbes (fixe, variable B) à partir d'une enzyme avec un ternaire-mécanisme complexe sont tracées dans une Lineweaver–Burk de la parcelle, l'ensemble des lignes de produits se croisent.
Les Enzymes avec ternaire-des mécanismes complexes qui comprennent de la glutathion S-transférase,[17] la dihydrofolate réductase[18] et de l'ADN polymérase.[19] Les liens suivants montrent de courtes animations du ternaire-mécanismes complexes de l'enzyme dihydrofolate réductase[β] et de l'ADN polymérase[γ].
Mécanisme "Ping–pong"[modifier | modifier le code]
Comme illustré sur la droite, les enzymes avec un mécanisme de ping-pong peut exister dans deux états, E et chimiquement modifié la forme de l'enzyme E*; cette enzyme modifiée est connu comme un intermédiaire. Dans de tels mécanismes, Un substrat se lie, les changements de l'enzyme de E*, par exemple, le transfert d'un groupe chimique pour le site actif, et est alors libérée. Seulement après le premier substrat est libéré peut substrat B lier et de réagir avec l'enzyme modifiée, la régénération de la non modifié E forme. Lorsqu'un ensemble de v [S] courbes (fixe, variable B) à partir d'une enzyme avec un mécanisme de ping–pong sont tracées dans une Lineweaver–Burk de la parcelle, un ensemble de lignes parallèles seront produites. Cela s'appelle une intrigue secondaire.
Les Enzymes de ping–pong mécanismes comprennent certains oxydoréductases tels que la thiorédoxine peroxydase,[20] transférases comme acylneuraminate cytidylyltransferase[21] et des sérine-protéases telles que la trypsine et la chymotrypsine.[22] les Sérine-protéases sont un très commune et diversifiée de la famille des enzymes, y compris digestive enzymes (trypsine, chymotrypsine, et de l'élastase), plusieurs enzymes de la cascade de la coagulation du sang et beaucoup d'autres. Dans ces protéases à sérine, l'E* intermédiaire est un acyl-enzyme espèces formées par l'attaque d'un site actif de la sérine résidu sur une liaison peptidique d'une protéine substrat. Un court métrage d'animation montrant le mécanisme de la chymotrypsine est lié ici.[δ]
Réversible, la catalyse et l'équation de Haldane[modifier | modifier le code]
Cinétique non Michaelienne[modifier | modifier le code]

Certaines enzymes de produire une sigmoïde v [S] de la parcelle, ce qui indique souvent la coopérative de liaison du substrat dans le site actif. Cela signifie que la liaison d'une molécule de substrat affecte la liaison de la suite des molécules de substrat. Ce comportement est plus fréquent dans multimère d'enzymes et de l'interaction de plusieurs sites actifs.[23] Ici, le mécanisme de coopération est similaire à celle de l'hémoglobine, avec la liaison du substrat à un site actif modifier l'affinité de l'autre des sites actifs de molécules de substrat. Une coopérativité Positive se produit lors de la liaison de la première molécule de substrat augmente l'affinité de l'autre des sites actifs de substrat. Coopérativité négative se produit lors de la liaison de la première substrat diminue l'affinité de l'enzyme pour les autres molécules de substrat.
Les enzymes allostériques inclure des mammifères tyrosyl arnt-synthétase, ce qui montre coopérativité négative,[24] et bactériennes de l'aspartate transcarbamoylase[25] et de la phosphofructokinase,[26] qui montrent une coopérativité positive.
La coopérativité est étonnamment commun et peut aider à réguler les réponses des enzymes à des changements dans les concentrations de leurs substrats. Une coopérativité Positive rend enzymes beaucoup plus sensible à [S] et leurs activités peuvent montrer de grands changements sur une gamme étroite de la concentration du substrat. À l'inverse, de coopérativité négative rend enzymes insensible à de petits changements dans [S].
La Colline de l'équation (biochimie)[27] est souvent utilisé pour décrire le degré de coopérativité quantitativement non-cinétique de Michaelis–Menten. Dérivé du coefficient de Hill n mesures combien la liaison du substrat à un site actif affecte la liaison du substrat pour les autres sites. Un coefficient de Hill de <1 indique une coopérativité négative et un coefficient supérieur à 1 indique positive de la coopérativité.
Cinétique pré-stationnaire[modifier | modifier le code]
Dans le premier moment, après une enzyme est mélangé avec le substrat, aucun produit n'a été formé et sans intermédiaires existent. L'étude de la prochaine quelques millisecondes de la réaction est appelée pré-état d'équilibre cinétique. Avant l'état d'équilibre cinétique est donc concerné par la formation et la consommation de l'enzyme–substrat intermédiaires (comme ES ou E*) jusqu'à ce que leur état d'équilibre, les concentrations sont atteintes.
Cette approche a d'abord été appliquée à la réaction d'hydrolyse catalysée par la chymotrypsine.[28] Souvent, la détection d'un intermédiaire est une pièce essentielle de la preuve dans les enquêtes de ce mécanisme d'une enzyme de la façon suivante. Par exemple, dans la table de ping–pong mécanismes qui sont indiqués ci-dessus, rapide des mesures cinétiques pouvez suivre le rejet d'un produit P et à mesure de la formation de l'enzyme modifiée intermédiaire E*.[29] Dans le cas de la chymotrypsine, cet intermédiaire est formé par une attaque sur le substrat par le nucléophile de la sérine du site actif et la formation de l'acyl-enzyme intermédiaire.
Dans la figure à droite, l'enzyme produit E* rapidement dans les premières secondes de la réaction. Le rythme ralentit, comme l'état d'équilibre est atteint. Ce rapide éclatement de la phase de la réaction des mesures d'un seul chiffre d'affaires de l'enzyme. Par conséquent, la quantité de produit libéré dans cet élan, montré que l'ordonnée à l'origine sur le y-axe du graphique, donne également le montant de la fonctionnelle de l'enzyme qui est présente dans le dosage.[30]
Mécanisme chimique[modifier | modifier le code]
Un objectif important de la mesure de la cinétique enzymatique est de déterminer le mécanisme chimique d'une réaction enzymatique, c'est à dire, la séquence d'étapes chimiques qui transforment substrat en produit. La cinétique des approches discutées ci-dessus va afficher à ce que les taux intermédiaires sont formés et inter-convertis, mais ils ne peuvent pas identifier exactement ce que ces intermédiaires sont.
Cinétique des mesures prises en vertu de diverses conditions de solution ou légèrement modifiés, des enzymes ou des substrats souvent faire la lumière sur ce mécanisme chimique, car ils révèlent le taux de la détermination de l'étape ou des intermédiaires de la réaction. Par exemple, la rupture d'une liaison covalente à un hydrogène, l'atome est un taux commun,-la détermination de l'étape. Les transferts d'hydrogène est le taux de la détermination peut être illustré par la mesure de la cinétique des effets de la substitution de chacun de l'hydrogène par le deutérium, de la stabilité de l'isotope. Le taux changera quand la critique de l'hydrogène est remplacé, en raison d'un principal effet isotopique cinétique, qui se produit en raison d'obligations de deutérium sont plus difficiles à briser que les obligations de l'hydrogène.[31] Il est également possible de mesurer des effets similaires avec d'autres isotopes des substitutions, comme 13C/12C et 18O/16O, mais ces effets sont de plus en plus subtiles.[32]
Les Isotopes peut également être utilisé pour révéler le sort des diverses parties de l'molécules de substrat dans les produits finaux. Par exemple, il est parfois difficile de discerner l'origine de l'oxygène de l'atome dans le produit final; car il peut provenir de l'eau ou de la partie du substrat. Ceci peut être déterminé par systématiquement la substitution de l'oxygène stable isotopes 18O dans les différentes molécules qui participent à la réaction et à la vérification de l'isotope dans le produit.[33] Le mécanisme chimique peut également être élucidée par l'examen de la cinétique et les effets isotopiques sous différentes conditions de pH,[34] en modifiant les ions métalliques ou d'autres liés cofacteurs,[35] par mutagenèse dirigée de conservé les résidus d'acides aminés, ou en étudiant le comportement de l'enzyme en présence d'analogues du substrat(s).[36]
Inhibition et activation[modifier | modifier le code]

Les inhibiteurs de l'Enzyme sont des molécules qui permettent de réduire ou d'abolir l'activité de l'enzyme, tandis que l'enzyme activateurs sont des molécules qui augmentent le taux catalytique des enzymes. Ces interactions peuvent être réversibles (c'est à dire, la suppression de l'inhibiteur de la rétablit l'activité de l'enzyme) ou irréversible (c'est à dire, l'inhibiteur de façon permanente inactive l'enzyme).
Les inhibiteurs réversibles[modifier | modifier le code]
Traditionnellement réversible inhibiteurs de l'enzyme ont été classés comme concurrentiels, non concurrentiels, ou de la non-concurrence, en fonction de leurs effets sur Km et Vmax. Ces différents effets de l'inhibiteur de la liaison à l'enzyme E, le complexe enzyme–substrat ES, ou les deux, respectivement. La division de ces classes se pose à partir d'un problème dans le calcul et les résultats dans la nécessité d'utiliser deux différentes constantes de liaison pour un événement de liaison. La liaison de l'inhibiteur et son effet sur l'activité enzymatique sont deux distinctement des choses différentes, un autre problème les équations traditionnelles ne parviennent pas à reconnaître. Dans inhibition non compétitive de la liaison de l'inhibiteur de résultats à 100% d'inhibition de l'enzyme seulement, et ne parvient pas à envisager la possibilité de quelque chose entre les deux.[37] Dans l'inhibition non compétitive, l'inhibiteur se lie à l'enzyme à son site allostérique; par conséquent, l'affinité de liaison, ou Km, du substrat à l'enzyme reste le même. D'autre part, la Vmax va diminuer par rapport à décomplexé de l'enzyme. Sur un Lineweaver-Burk de la parcelle, la présence d'un inhibiteur non compétitif est illustré par un changement dans l'ordonnée à l'origine, défini comme 1/Vmax. Le x-intercepter, définie comme -1/Km, restera le même. Dans inhibition compétitive, l'inhibiteur se lie à l'enzyme sur le site actif, en compétition avec le substrat. En conséquence, les Km vont augmenter et la Vmax reste le même.[38] La forme la plus commune de l'inhibiteur terme obscurcit la relation entre l'inhibiteur de la liaison à l'enzyme et de son rapport à toute autre liaison terme soit l'équation de Michaelis–Menten ou une courbe dose-réponse associé à un ligand de récepteur de liaison. Pour démontrer la relation suivante réarrangement peut être faite:
L'ajout de zéro vers le bas ([I]-[I])
En divisant par [I]+Kje
Cette notation montre que similaire à l'équation de Michaelis–Menten, où la vitesse de la réaction dépend de la pour cent de l'enzyme de la population en interaction avec le substratModèle:Fragment
fraction de l'enzyme de la population lié par le substrat
fraction de l'enzyme de la population lié par un inhibiteur de
l'effet de l'inhibiteur est un résultat de la pour cent de l'enzyme de la population en interaction avec l'inhibiteur. Le seul problème avec cette équation sous sa forme actuelle, est qu'il suppose absolue de l'inhibition de l'enzyme avec l'inhibiteur de liaison, alors qu'en fait, il peut y avoir un large éventail d'effets n'importe où à partir de 100% d'inhibition de substrat au juste >0%. Pour tenir compte de cette équation peut être facilement modifié pour tenir compte des différents degrés de l'inhibition par l', y compris un delta Vmax terme.
ou
Ce terme peut alors définir l'activité enzymatique résiduelle présente lorsque l'inhibiteur est en interaction avec les enzymes individuels dans la population. Toutefois, l'inclusion de ce terme est la valeur ajoutée de la possibilité d'activation si le secondaire Vmax terme s'avère être supérieure à la durée initiale. Pour tenir compte de la possibilité d'activation ainsi que la notation peut alors être réécrite en remplacement de l'inhibiteur de "je" avec un modificateur terme notée ici en tant que "X".
Bien que cette terminologie résultats de simplifier la façon de les traiter avec des effets cinétiques relatives à la vitesse maximale de l'équation de Michaelis–Menten, il met en évidence les problèmes potentiels avec le terme utilisé pour décrire les effets relatifs à la Km. Le Km concernant à l'affinité de l'enzyme pour le substrat, dans la plupart des cas sont liés à des changements potentiels dans le site de liaison de l'enzyme qui serait directement résulter de l'inhibiteur de l'enzyme de interactions. En tant que tel une durée similaire à celle proposée ci-dessus pour moduler Vmax doit être appropriée dans la plupart des situations:[39][40]
Les inhibiteurs irréversibles[modifier | modifier le code]
Les inhibiteurs de l'Enzyme peut aussi de façon irréversible à inactiver les enzymes, généralement par une liaison covalente modification des résidus du site actif. Ces réactions, qui peut être appelé le suicide des substrats, suivez décroissance exponentielle des fonctions et sont généralement saturable. Au-dessous de la saturation, ils suivent de premier ordre de la cinétique à l'égard de l'inhibiteur.
Les mécanismes de la catalyse[modifier | modifier le code]

L'favorisée modèle pour l'enzyme–substrat interaction est l'ajustement induit modèle.[41] Ce modèle propose que l'interaction initiale entre l'enzyme et le substrat est relativement faible, mais que ces interactions faibles de provoquer rapidement des changements de conformation de l'enzyme de renforcer la liaison. Ces conformation des changements aussi apporter des résidus catalytiques dans le site actif de près de liaisons chimiques dans le substrat qui sera modifié dans la réaction.[42] les changements de Conformation peut être mesurée à l'aide de dichroïsme circulaire ou à double polarisation de l'interférométrie. Après la liaison a lieu, un ou plusieurs mécanismes de la catalyse diminuer l'énergie de la réaction de l'état de transition en offrant une autre voie chimique de la réaction. Les mécanismes de la catalyse comprennent la catalyse par l'émission d'obligations de la souche; par la proximité et de l'orientation; par le site actif de protons donneurs ou accepteurs; covalente de la catalyse et de creusement de tunnel quantique.[43]
La cinétique enzymatique ne peut pas prouver quels sont les modes de la catalyse sont utilisés par une enzyme. Cependant, certaines données cinétiques peuvent suggérer des possibilités d'être examinées par d'autres techniques. Par exemple, un mécanisme de ping–pong avec l'éclatement de la phase de pré-état d'équilibre cinétique suggère la catalyse covalente peut être important dans cette enzyme mécanisme. Sinon, l'observation d'un fort effet du pH sur la Vmax mais pas Km pourrait indiquer que les résidus du site actif doit être en particulier un détecteur à ionisation de l'état pour la catalyse de se produire.
Aspects historiques[modifier | modifier le code]
En 1902, Victor Henri proposé une théorie quantitative de la cinétique enzymatique,[44], mais au moment de l'expérimental, de l'importance de la concentration en ions hydrogène n'était pas encore reconnue. Après Peter Lauritz Sørensen a défini les logarithmique pH-échelle et introduit le concept de mise en mémoire tampon en 1909[45] le chimiste allemand Leonor Michaelis et le Dr Maud Leonora Menten (un chercheur post-doctoral dans Michaelis du laboratoire à l'époque)répété Henri expériences et confirmé son équation, qui est maintenant généralement dénommé cinétique de Michaelis-Menten (parfois aussi Henri-cinétique de Michaelis-Menten).[46] Leur travail a été développé par le G. E. Briggs et J. B. S. Haldane, qui dérivées des équations cinétiques qui sont encore largement considéré aujourd'hui un point de départ dans la modélisation de l'activité enzymatique.[47]
La contribution majeure de Henri-Michaelis-Menten approche a été de penser à des réactions enzymatiques en deux étapes. Dans la première, le substrat se lie de façon réversible à l'enzyme, formant le complexe enzyme-substrat. Cela est parfois appelé le Michaelis complexe. L'enzyme puis catalyse l'étape chimique de la réaction et libère le produit. La cinétique de nombreuses enzymes est bien décrit par le simple Michaelis-Menten modèle, mais toutes les enzymes ont des mouvements internes qui ne sont pas pris en compte dans le modèle et peut avoir d'importantes contributions à l'ensemble de la cinétique de la réaction. Cela peut être modélisé par l'introduction de plusieurs de Michaelis-Menten voies qui sont liés à la fluctuation des taux,[48][49][50] qui est une mathématique de l'extension de la base de Michaelis Menten mécanisme.[51]
Logiciel[modifier | modifier le code]
Références[modifier | modifier le code]
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Enzyme kinetics » (voir la liste des auteurs).
- Wrighton, Mark S. et Ebbing, Darrell D., General chemistry, Boston, 4th, (ISBN 0-395-63696-5)
- Fromm H.J., Hargrove M.S. (2012) Enzyme Kinetics. In: Essentials of Biochemistry. Springer, Berlin, Heidelberg
- Danson, Michael et Eisenthal, Robert, Enzyme assays: a practical approach, Oxford [Oxfordshire], Oxford University Press, (ISBN 0-19-963820-9)
- « Single-molecule enzymology », J. Biol. Chem., vol. 274, no 23, , p. 15967–70 (PMID 10347141, DOI 10.1074/jbc.274.23.15967)
- Lu H, « Single-molecule spectroscopy studies of conformational change dynamics in enzymatic reactions », Current pharmaceutical biotechnology, vol. 5, no 3, , p. 261–9 (PMID 15180547, DOI 10.2174/1389201043376887)
- « Structure, dynamics, and catalytic function of dihydrofolate reductase », Annual Review of Biophysics and Biomolecular Structure, vol. 33, , p. 119–40 (PMID 15139807, DOI 10.1146/annurev.biophys.33.110502.133613)
- Duggleby RG, « Analysis of enzyme progress curves by non-linear regression », Methods in Enzymology, vol. 249, , p. 61–90 (ISBN 978-0-12-182150-0, PMID 7791628, DOI 10.1016/0076-6879(95)49031-0)
- « A pH-dependent conformational change, rather than the chemical step, appears to be rate-limiting in the hammerhead ribozyme cleavage reaction », J. Mol. Biol., vol. 315, no 2, , p. 121–30 (PMID 11779233, DOI 10.1006/jmbi.2001.5145)
- 108 – 1010 M−1 s−1
- « The moderately efficient enzyme: evolutionary and physicochemical trends shaping enzyme parameters », Biochemistry, vol. 50, no 21, , p. 4402–10 (PMID 21506553, DOI 10.1021/bi2002289)
- Jones ME, « Analysis of algebraic weighted least-squares estimators for enzyme parameters », Biochem. J., vol. 288, no Pt 2, , p. 533–8 (PMID 1463456, PMCID 1132043, lire en ligne [PDF])
- Jones ME, « A comparison of the parameter estimating procedures for the Michaelis-Menten model », J. Theor. Biol., vol. 145, no 4, , p. 457–64 (PMID 2246896, PMCID 1132043, DOI 10.1016/S0022-5193(05)80481-3)
- « A normalized plot as a novel and time-saving tool in complex enzyme kinetic analysis », Biochem. J., vol. 358, no Pt 3, , p. 573–83 (PMID 11577687, PMCID 1222113, lire en ligne)
- « Global organization of metabolic fluxes in the bacterium Escherichia coli », Nature, vol. 427, no 6977, , p. 839–43 (PMID 14985762, PMCID 1222113, DOI 10.1038/nature02289, Bibcode 2004Natur.427..839A, arXiv q-bio/0403001, lire en ligne)
- « An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR) », Genome Biol., vol. 4, no 9, , R54 (PMID 12952533, PMCID 193654, DOI 10.1186/gb-2003-4-9-r54)
- for a complete derivation, see here
- « X-ray crystal structures of cytosolic glutathione S-transferases. Implications for protein architecture, substrate recognition and catalytic function », Eur. J. Biochem., vol. 220, no 3, , p. 645–61 (PMID 8143720, DOI 10.1111/j.1432-1033.1994.tb18666.x)
- « Dihydrofolate reductase from Escherichia coli: the kinetic mechanism with NADPH and reduced acetylpyridine adenine dinucleotide phosphate as substrates », Biochemistry, vol. 27, no 15, , p. 5493–9 (PMID 3052577, DOI 10.1021/bi00415a016)
- Fisher PA, « Enzymologic mechanism of replicative DNA polymerases in higher eukaryotes », Progress in Nucleic Acid Research and Molecular Biology, vol. 47, no 15, , p. 371–97 (ISBN 978-0-12-540047-3, PMID 8016325, DOI 10.1016/S0079-6603(08)60257-3)
- « 2-Cys peroxiredoxin PfTrx-Px1 is involved in the antioxidant defence of Plasmodium falciparum », Mol. Biochem. Parasitol., vol. 130, no 2, , p. 75–81 (PMID 12946843, DOI 10.1016/S0166-6851(03)00161-0)
- « Kinetic properties of the acylneuraminate cytidylyltransferase from Pasteurella haemolytica A2 », Biochem. J., vol. 358, no Pt 3, , p. 585–98 (PMID 11577688, PMCID 1222114, DOI 10.1016/S0166-6851(03)00161-0, lire en ligne)
- Kraut J, « Serine proteases: structure and mechanism of catalysis », Annu. Rev. Biochem., vol. 46, no Pt 3, , p. 331–58 (PMID 332063, PMCID 1222114, DOI 10.1146/annurev.bi.46.070177.001555, lire en ligne)
- « Co-operative and allosteric enzymes: 20 years on », Eur. J. Biochem., vol. 166, no 2, , p. 255–72 (PMID 3301336, DOI 10.1111/j.1432-1033.1987.tb13510.x)
- « Tyrosyl-tRNA synthetase acts as an asymmetric dimer in charging tRNA. A rationale for half-of-the-sites activity », Biochemistry, vol. 27, no 15, , p. 5525–30 (PMID 3179266, DOI 10.1021/bi00415a021)
- « Allosteric Regulation of Catalytic Activity: Escherichia coli Aspartate Transcarbamoylase versus Yeast Chorismate Mutase », Microbiol. Mol. Biol. Rev., vol. 65, no 3, , p. 404–21, table of contents (PMID 11528003, PMCID 99034, DOI 10.1128/MMBR.65.3.404-421.2001, lire en ligne)
- « Structural basis of the allosteric behaviour of phosphofructokinase », Nature, vol. 343, no 6254, , p. 140–5 (PMID 2136935, PMCID 99034, DOI 10.1038/343140a0, Bibcode 1990Natur.343..140S, lire en ligne)
- Hill, A. V. The possible effects of the aggregation of the molecules of haemoglobin on its dissociation curves. J. Physiol., 1910 40, iv–vii.
- « The reaction of p-nitrophenyl esters with chymotrypsin and insulin », Biochem. J., vol. 56, no 2, , p. 288–97 (PMID 13140189, PMCID 1269615)
- Fersht, Alan, « Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding », Biochem. J., San Francisco, W.H. Freeman, vol. 56, no 2, , p. 288–97 (ISBN 0-7167-3268-8, PMID 13140189, PMCID 1269615)
- « The determination of the concentration of hydrolytic enzyme solutions: alpha-chymotrypsin, trypsin, papain, elastase, subtilisin, and acetylcholinesterase », J. Am. Chem. Soc., vol. 88, no 24, , p. 5890–913 (PMID 5980876, DOI 10.1021/ja00976a034)
- Cleland WW, « The use of isotope effects to determine enzyme mechanisms », Arch. Biochem. Biophys., vol. 433, no 1, , p. 2–12 (PMID 15581561, DOI 10.1016/j.abb.2004.08.027)
- Northrop D, « The expression of isotope effects on enzyme-catalyzed reactions », Annu. Rev. Biochem., vol. 50, , p. 103–31 (PMID 7023356, DOI 10.1146/annurev.bi.50.070181.000535)
- « Drug biotransformation: mechanistic studies with stable isotopes », Journal of clinical pharmacology, vol. 26, no 6, , p. 448–51 (PMID 3734135, DOI 10.1002/j.1552-4604.1986.tb03556.x)
- Cleland WW, « Use of isotope effects to elucidate enzyme mechanisms », CRC Crit. Rev. Biochem., vol. 13, no 4, , p. 385–428 (PMID 6759038, DOI 10.3109/10409238209108715)
- « Catalysis by metal-activated hydroxide in zinc and manganese metalloenzymes », Annu. Rev. Biochem., vol. 68, , p. 33–57 (PMID 10872443, DOI 10.1146/annurev.biochem.68.1.33)
- « Challenges in enzyme mechanism and energetics », Annu. Rev. Biochem., vol. 72, , p. 517–71 (PMID 12704087, DOI 10.1146/annurev.biochem.72.121801.161617)
- R. Walsh, E. Martin et S. Darvesh, « Limitations of conventional inhibitor classifications », Integrative Biology, Royal Society of Chemistry, vol. 3, no 12, , p. 1197–1201 (PMID 22038120, DOI 10.1039/c1ib00053e)
- W.W Cleland, « The kinetics of enzyme-catalyzed reactions with two or more substrates or products: III. Prediction of Initial Velocity and Inhibition Patterns by Inspection », Elsevier, vol. 67, {{Article}} : paramètre «
date» manquant, p. 188–196 (DOI 10.1016/0006-3002(63)91816-x) - R. Walsh, E. Martin et S. Darvesh, « A versatile equation to describe reversible enzyme inhibition and activation kinetics: Modeling β-galactosidase and butyrylcholinesterase », Biochimica et Biophysica Acta (BBA) - General Subjects, vol. 1770, no 5, , p. 733–746 (PMID 17307293, DOI 10.1016/j.bbagen.2007.01.001)
- Walsh, Ryan, Medicinal Chemistry and Drug Design, InTech, , 357–371 p. (ISBN 978-953-51-0513-8, lire en ligne), « Ch. 17. Alternative Perspectives of Enzyme Kinetic Modeling »
- Koshland DE, « Application of a Theory of Enzyme Specificity to Protein Synthesis », Proc. Natl. Acad. Sci. U.S.A., vol. 44, no 2, , p. 98–104 (PMID 16590179, PMCID 335371, DOI 10.1073/pnas.44.2.98, Bibcode 1958PNAS...44...98K)
- Hammes G, « Multiple conformational changes in enzyme catalysis », Biochemistry, vol. 41, no 26, , p. 8221–8 (PMID 12081470, DOI 10.1021/bi0260839)
- « A new conceptual framework for enzyme catalysis. Hydrogen tunnelling coupled to enzyme dynamics in flavoprotein and quinoprotein enzymes », Eur. J. Biochem., vol. 269, no 13, , p. 3096–102 (PMID 12084049, DOI 10.1046/j.1432-1033.2002.03020.x, lire en ligne)
- Walsh, Ryan, Theorie generale de l'action de quelques diastases, vol. 135, InTech, , 916–9 p. (ISBN 978-953-51-0513-8, lire en ligne), « Ch. 17. Alternative Perspectives of Enzyme Kinetic Modeling »
- (de) « Enzymstudien {II}. Über die Messung und Bedeutung der Wasserstoffionenkonzentration bei enzymatischen Prozessen », Biochem. Z., vol. 21, , p. 131–304
- (de) « Die Kinetik der Invertinwirkung », Biochem. Z., vol. 49, , p. 333–369; « The original Michaelis constant: translation of the 1913 Michaelis-Menten paper », Biochemistry, vol. 50, no 39, , p. 8264–9 (PMID 21888353, PMCID 3381512, DOI 10.1021/bi201284u)
- « A Note on the Kinetics of Enzyme Action », The Biochemical Journal, vol. 19, no 2, , p. 339–339 (PMID 16743508, PMCID 1259181, DOI 10.1042/bj0190338)
- « Stretched exponential decay and correlations in the catalytic activity of fluctuating single lipase molecules », Proceedings of the National Academy of Sciences of the United States of America, vol. 102, no 7, , p. 2368–2372 (PMID 15695587, PMCID 548972, DOI 10.1073/pnas.0409039102, Bibcode 2005PNAS..102.2368F)
- « Ever-fluctuating single enzyme molecules: Michaelis-Menten equation revisited », Nature Chemical Biology, vol. 2, no 2, , p. 87–94 (PMID 16415859, DOI 10.1038/nchembio759)
- « Single-molecule enzymatic dynamics », Science, vol. 282, no 5395, , p. 1877–1882 (PMID 9836635, DOI 10.1126/science.282.5395.1877)
- « Single molecule Michaelis-Menten equation beyond quasistatic disorder », Physical Review E, vol. 74, no 3 Pt 1, , p. 030902 (PMID 17025584, DOI 10.1103/PhysRevE.74.030902, Bibcode 2006PhRvE..74c0902X, arXiv cond-mat/0604364)
[[Catégorie:Catalyse]]



